单细胞mRNA测序(scRNA-seq)允许分析异质细胞群,提供解决各种生物和医学问题的令人兴奋的可能性。近期新开发了一系列方法,因此有必要系统地比较它们的灵敏度,准确度,精度和成本效率。
在这里,来自Ludwig-Maximilians大学的研究人员已经生成并分析了来自479只小鼠ES细胞和加标对照的scRNA-seq数据,这些对照用四种不同方法制备,每种方法分别进行两次重复。他们通过检测基因的数量和它们捕获加标mRNA的效率,通过将加标mRNA浓度与估计的表达水平相关联,通过功率模拟和方差分解的准确度以及它们的效率来比较它们的灵敏度。达到一定数量电力的成本。
实验和计算管道的示意图
在2i / LIF和ERCC加标RNA中培养的小鼠胚胎干细胞用于制备单细胞RNA-seq文库。这四种方法的不同之处在于独特的分子标识符序列(UMI)的存在和长度,允许鉴定cDNA扩增过程中产生的读数。对于所有方法,比较地进行细胞的数据处理和子集化。每个方法和重复的应用,使其细胞数用它们的平均测序深度显示。颜色代表比较的scRNA-seq方法:紫色 - CEL-seq,橙色 - Drop-seq,绿色SCRB-seq,蓝色 - Smart-seq并且在本研究中使用。
虽然所有方法的准确度相似,但实验研究人员发现微流体平台上的Smart-seq是较敏感的方法,CEL-seq是较准确的方法,SCRB-seq和Drop-seq是比较有效的方法。该分析为四种可用的scRNA-seq方法的选择提供了坚实的基础,并为未来的方法开发提供了基准。
scRNA-seq方法的灵敏度
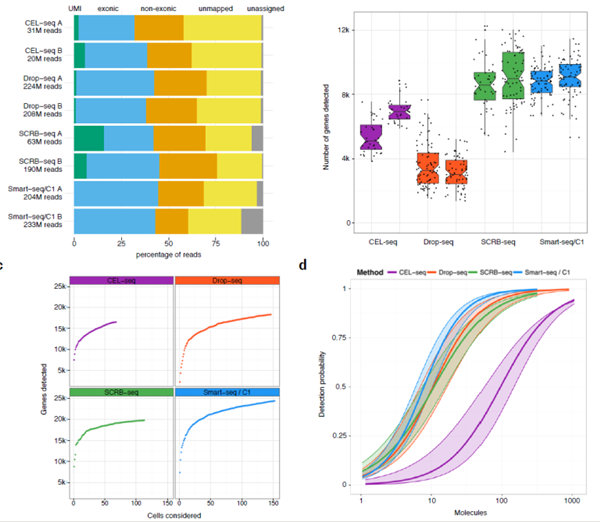
(a)不能分配给细胞条形码(灰色)的总读数百分比,不能映射到小鼠基因组(黄色),
映射到外显子(橙色)外部区域,内部外显子(蓝色)和携带独特的UMI(绿色)。(b)总共读数超过100,000的所有细胞中至少有一次读数的基因数。每个点代表一个细胞,每个箱图代表每个重复和方法的中位数。(c)添加更多细胞时检测到的累积基因数。(d)每种方法的灵敏度估计为检测ERCC转录物的概率取决于其每个细胞的拷贝数。估计值的95%置信区间显示为阴影区域。