哈佛大学的两个团队将微流体技术引入单细胞RNA-Seq方法中,分别开发出Drop-seq和inDROP。2015年,这两种方法发表在同一期的《Cell》杂志上。它们的出现,让快速、低成本地分析数千个单细胞的基因活性成为现实。
众所周知,大脑是一种复杂的结构。它包括近860亿个神经元以及数十亿个其他类型的细胞。单细胞的基因表达分析能够揭开细胞之间的差异,帮助绘制复杂组织的细胞多样性图谱。不过,如果每次只分析一个细胞,那显然不现实。
于是,哈佛大学的两个团队将微流体技术引入单细胞RNA-Seq方法中,分别开发出Drop-seq和inDROP。2015年,这两种方法发表在同一期的《Cell》杂志上。它们的出现,让快速、低成本地分析数千个单细胞的基因活性成为现实。
这两种技术都利用微流体装置将带有条形码的微珠和细胞一起装入微小的液滴。这些液滴在一个小型设备上生成,沿着一根头发宽的槽道流动。条形码附着到每个细胞的一些基因上,因此科学家们可以一次测序所有的基因,追踪每个基因的来源细胞。
Drop-seq
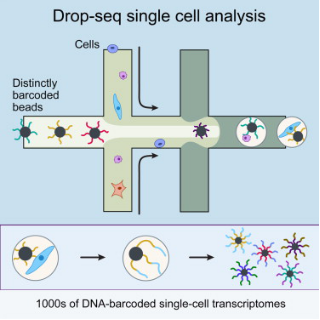
Drop-seq由哈佛医学院Steven McCarroll领导的团队开发。利用这种方法,他们已经分析了来自小鼠眼睛视网膜的44,800多个细胞的基因活性,鉴定出39种不同类型的视网膜细胞。他们的下一步目标是构建大脑的整体结构,以便对参与正常神经发育的基因有一个清晰的了解。
这些雄心勃勃的研究项目之所以能够实施,是因为Drop-seq的成本更低,速度更快。据介绍,利用Drop-seq,分析单细胞的基因活性仅需7美分。此外,分析过程也相当高效,每个科学家每天可以分析约1万个细胞。
inDrop
inDrop方法是由哈佛大学的系统生物学教授Marc Kirschner领导开发的,与Drop-seq有些类似,但不完全相同。研究小组利用这种方法来分析小鼠的数千个胚胎干细胞和分化细胞,以便更好地了解干细胞分化。
Drop-seq的微珠库中有1600万个条形码,而inDrop方法只产生约15万个条形码,这意味着它每次运行处理的细胞数较少。不过,当实验研究人员想分析少量的组织样本时,inDrop可能更有优势。因为它捕获细胞的比例高于Drop-seq。

测序技术大比拼
在各种单细胞RNA测序技术被陆续开发出以后,相信大家的选择困难综合征又犯了,不知道该如何选择。好在,德国慕尼黑大学的研究人员比较了几种常用的几种方法,包括Smart-Seq、CEL-Seq、SCRB-seq以及Drop-seq。他们生成并分析了479个小鼠胚胎干细胞的RNA-Seq数据。
研究人员发现,尽管所有方法的准确性相似,但是Smart-seq的灵敏度较高,CEL-seq的数据较准确,而SCRB-seq和Drop-seq的应用效率较为。有了这个分析,相信你在选择方法时心里有底了吧。