细胞是生物结构和功能的基本单位,种类和状态差异很大。在大多数生物系统中,我们对细胞多样性的认识不完整,对于像神经系统(脑细胞)这样的复杂组织 。单细胞身份和功能的表征,作为对每个细胞功能能力和反应的理解细胞类型,将加速生物学发现。它可能用于癌症,用于肿瘤,几乎任何可能在细胞群体中具有多样性的物体。然而,今天的技术并没有提供足够简单的方法来同时分析大量的单个细胞。
这样,需要快速,可扩展的方法来表征具有许多细胞类型和状态的复杂组织。
DROP-SEQ的原则和优点:
Drop-Seq是基于使用微流控技术快速分析数千个单个细胞的策略,通过将它们封装在微滴中进行并行分析。
这些纳升级水相室已被用于微流体装置中的许多应用:纳米制造,乳液和泡沫,药物递送,但也作为用于PCR 和逆转录的微小反应室 。Drop-Seq使用液滴将细胞划分为纳升级大小的反应室,用于分析它们的mRNA转录物,同时使用分子条形码策略记住转录物的起源细胞。利用这种技术,一位科学家每天可以同时制作10 000个单细胞文库,同时进行廉价且简单的实验。因此该方法将允许为已知的细胞类别和新的候选细胞亚型创建基因表达的分子图谱。
Drop-seq利用微流体的优势在于:
● 高吞吐量在短时间内产生;
● 尽量减少昂贵样品的消耗;
DROP-SEQ包含以下步骤:
● 从组织中制备单细胞悬液
● 准备条形码的引物(无论是在微粒的表面,内部)
● 使用微流体装置将每个细胞分别与微滴中的明显条形码化的微粒共同封装
● 一旦在液滴中分离,裂解细胞,释放它们的mRNA,然后与引物杂交
● 打破水滴并产生STAMPs(附着于微粒的单细胞转录组)
● 放大STAMP
● 测序和分析:使用STAMP条形码来推断每个转录本的原始细胞
Drop-Seq可以使用两种类型的珠子:
A. “简单”微粒
B. 水凝胶微粒
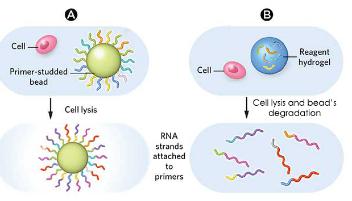
DROP-SEQ使用简单的微粒
1.从复杂的组织中制备单细胞悬液
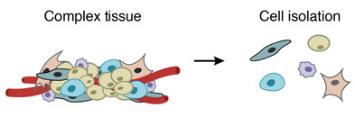
一个复杂的组织被分解成单个细胞。
2.引物合成
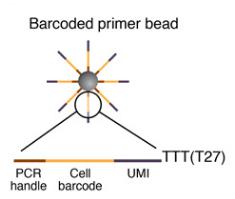
微粒上的引物序列
每个微粒含有超过10 的8次方个独立的引物,它们共享相同的“PCR处理”和“细胞条形码”,但具有不同的独特分子标识符(UMI)。实际上,PCR处理是在所有引物和珠子上相同的恒定序列,其允许STAMP形成后的PCR扩增。细胞条码只在相同微粒的所有引物中相同,但与其他珠子上的细胞条码不同,允许恢复细胞的来源。每个引物上不同的UMI允许mRNA转录物进行数字计数并识别PCR重复。在所有引物序列的末端存在30-bp oligodT序列以捕获mRNA并启动逆转录。
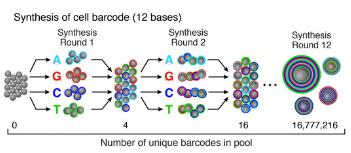
合成一个独特的分子标识符(UMI)
UMIs的合成发生在“分裂和合并”合成周期完成之后。所有微粒一起进行八轮简并合成,每个循环期间可用全部四种DNA碱基,使得每个单独的引物接收4,8 (65,536)个可能序列(UMI)中的一个。
3.微流体装置
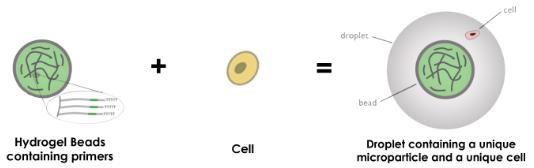
一旦单细胞悬液和微粒准备就绪后,使用定制设计的微流体装置将单个细胞与微粒一起封装在液滴中。
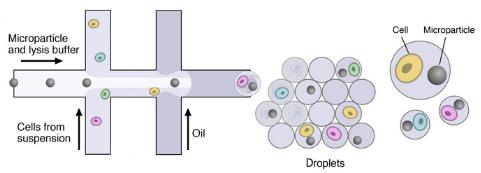
该装置在将它们分隔成不连续的液滴之前将两个水流连接起来。层流防止在液滴形成之前混合两种含水输入物。一个流体包含细胞,另一个流体包含悬浮在裂解缓冲液中的条形码化的引物珠粒。
微流体设置
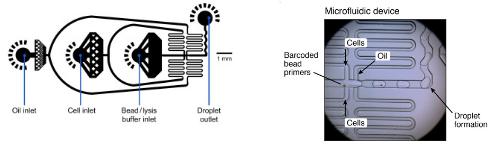
用于Drop-Seq的微流体装置的示例
流量
油:15,000μl/小时=250μl/分钟
细胞:4,000μl/ hr?66,7μl/ min
珠粒:4,000μl/ hr?66,7μl/ min
组件列表
§ 倒置显微镜
§ 压力控制器+ 3流量传感器/三个注射泵
§ 3个falcon管/ 3mL注射器
§ 磁力搅拌系统
§ 微流体管用于设置元件的流体连接§ 微流体配件和连接器
§ PDMS共流微流体微滴生成装置
§ 100微米细胞过滤器的珠子
§ 40微米细胞过滤器的细胞
§ 计数室
4.细胞裂解和RNA杂交
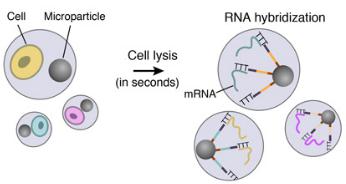
紧接着液滴形成后,每个细胞在液滴内裂解并且其mRNA释放。然后它们与其伴侣微粒表面上的引物杂交。
5. STAMPS一代
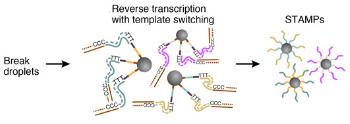
为了一次有效地生成数千个STAMP,通过添加试剂来破坏液滴以使油 - 水界面不稳定,并且收集微粒并洗涤。然后将mRNA在一个反应中一起逆转录成cDNA,形成称为“附着于微粒的单细胞转录组”的(STAMP)的一组珠子。
6. STAMPS的放大
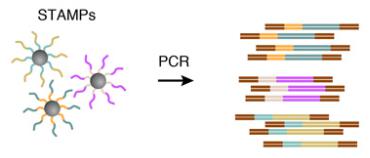
然后可以通过PCR反应对带有条形码的STAMP进行扩增以进行高通量mRNA测序,以分析任何期望数量的单个细胞。
7.测序和分析
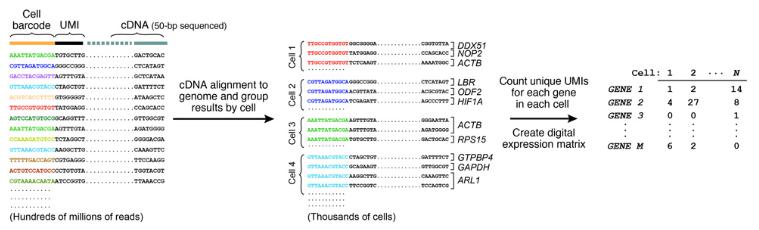
使用高容量并行测序从每个末端对得到的分子进行测序。读数首先与参考基因组比对以鉴定cDNA的来源基因。接下来,通过它们的细胞条形码来组织阅读,并且每个细胞中确定的每个基因的mRNA转录物的数量被数字计数。这就是UMI起作用的地方,避免从相同的mRNA转录物中重复计数序列读数。然后可以建立数字基因表达测量矩阵(每个基因每个细胞一个测量)用于进一步分析。
液滴下使用水凝胶微粒
关于使用水凝胶珠,操作原理或多或少保持不变,它们之间的区别涉及引物,它们位于微粒内部而不是在其表面。
为此,微流体装置由三个通道而不是两个组成:
§ 一个带来珠(这整个过程的关键)
§ 将细胞带入液滴中的一种
§ 一个带来我们需要进行分析的化学试剂
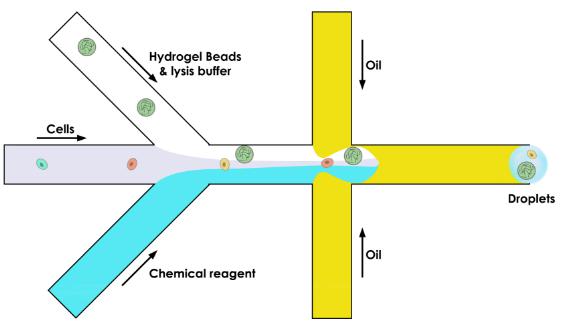
至于“简单微粒”的使用,水凝胶珠含有可用于逆转录反应的引物,其随后将条形码液滴内细胞的内容物条码化。因此获得了作为RNA序列拷贝的DNA序列集合,并且这些序列现在通过它们来自何处而被分类。
然后,有可能破坏液滴并将整个细胞群作为批量样品处理,因为知道每个细胞都被单独条形码化。
汶颢提供微液滴量产设备、液滴发生芯片、夹具、收集装置等。
文章内容摘自网络提供,如违反,请告知。
上一篇:利用Drop-seq单细胞测序技术绘制出复杂动物涡虫的
下一篇:Drop-seq