1. 为什么要做单细胞RNA测序(ScRNA-seq)?
主要解决的问题主要有两个:一是异质性(heterogeneity),比如a)鉴定某些因为含量(比例)较低而在常规RNA测序检测时被“掩盖”的细胞亚群;b) 检测细胞群体中不同的个体细胞:比如表达不同TCR的T细胞,胚胎发育早期的各个细胞等;c)追踪某群细胞内细胞间的谱系(lineage)或发育关系;二是获得基因表达、剪接等信息以及根据这些信息构建的调控关系。
2. 做单细胞测序的基本步骤是什么?
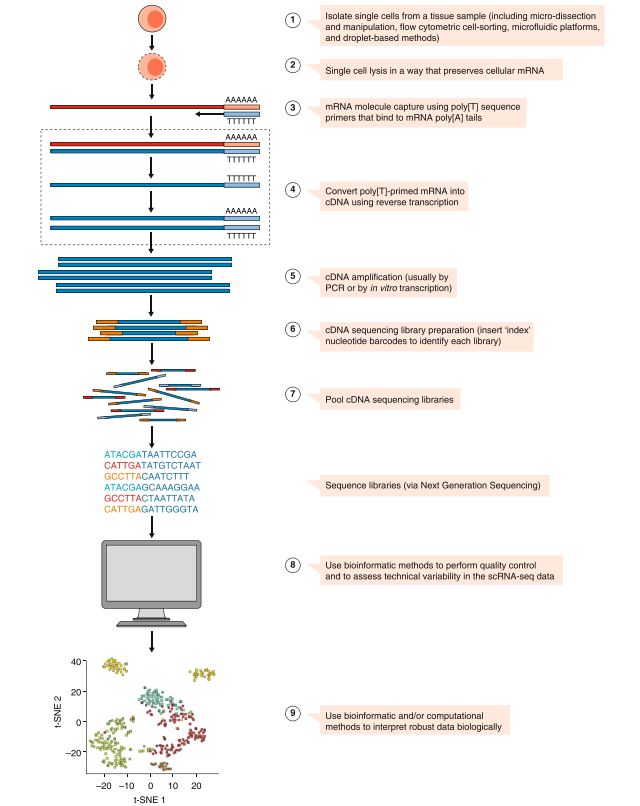
如上图所示,共分为9个步骤:
1)单细胞分离;
2) 保留mRNA的细胞裂解;
3) mRNA捕获;
4)RNA反转成cDNA;
5)cDNA扩增;
6)cDNA测序文库制备;
7)序列文库混样(pooling);
8)生物信息学工具进行质控;
9)专业的工具进行分析和结果展示;
3. 单细胞测序检测的样品类型有哪些?
理论上说,任何的真核细胞都可以进行scRNA-seq检测。但在实际操作过程中,需要注意的是:1)从组织(特别是实体组织)中获得细胞的数量和活性;2) 在获取过程中人为操作对细胞转录组的影响,以避免出现人为因素的转录表达变化。当然从技术上说,除了分离单个细胞进行检测外,还可以直接分离细胞核(nuclei)进行测序。
4. 单细胞测序的方法选择?
总体上有大约20种protocol,各个protocol的比较如下图所示:
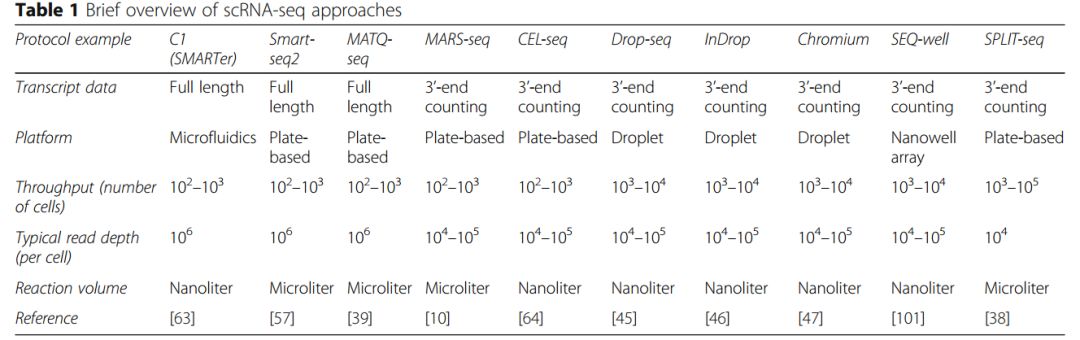
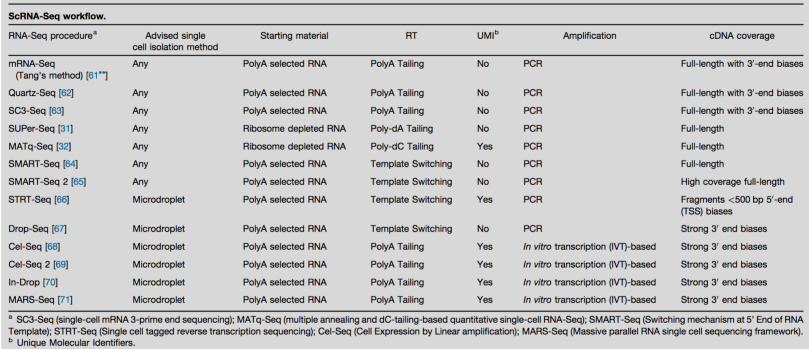
我们可以看到转录本的长度包括了:全长(Full length)和3’端测序,需要注意的是对于表达量较低的RNA来说更建议全长测序。其它的信息还包括了测序的平台(Droplet、Nanowell等)、测序通量(细胞的数量)、测序深度、反应体系和参考文献等。
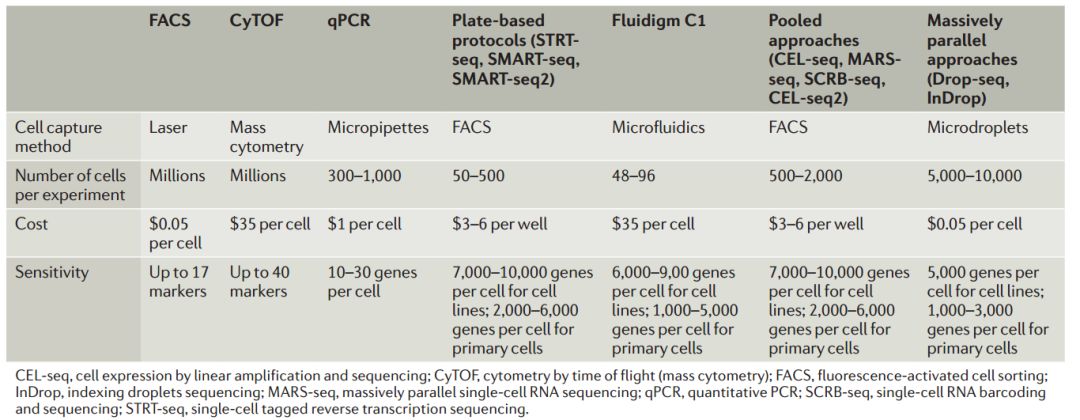
5. 需要测定的细胞数量以及测序深度该如何确定?
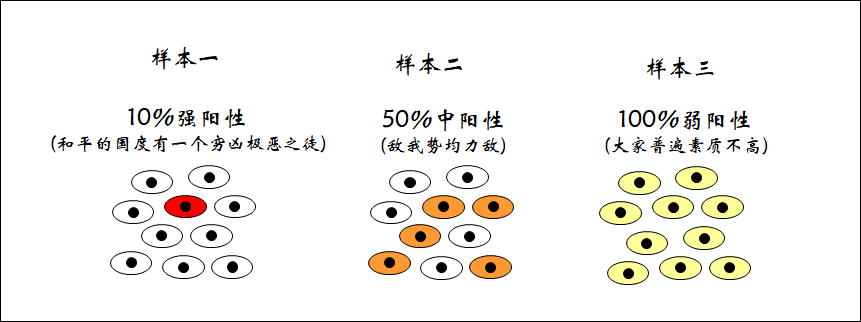
总体上说,大家需要考虑的因素包括研究目的、细胞的异质性大小、平台和价格。一般来说,异质性越高,我们需要检测的细胞亚群比例越低,需要测定的细胞数量就越多,大家可以类比考虑,比如A袋子有5个红球、5个蓝球组成;B袋子有赤橙黄绿青蓝紫7个颜色组成的10个球,其中我们关心的蓝球只有1个,如果我们想抽到篮球,是不是要抽更多次B袋子?

6. 单细胞RNA测序与常规RNA测序的区别是什么?
主要有三点:
1) 单细胞测序中某些低风度的转录本不能被检测到;
2)单细胞测序比常规测序的会有更高的技术误差(包括检测噪音等);
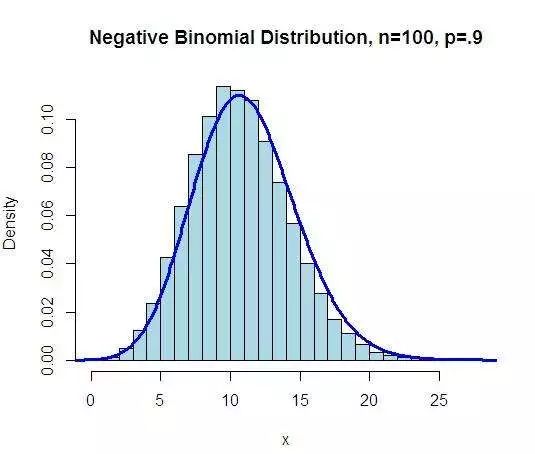
3)单细胞测序检测的转录本量的分布上更加复杂,一般是符合负二项分布的,但也出现其它的分布,因此在选择统计方法时一定要特别注意。
7. 单细胞RNA测序的分析该如何做?
这一部分我们下次单独写一期文章来介绍。
8. 单细胞RNA测序未来5年的前景如何?
主要包括几点:
1) 对检测细胞(样本)的要求降低:从新鲜细胞到冷冻保存的细胞;
2)性价比的提高:包括了平台技术的进步导致价格进一步降低和检测通量的增加:
3)对低丰度RNA检测的问题;
4)新的生信工具和软件的出现:后面我们会为大家介绍一个数据库——SCPortalen,来看别人的单细胞测序技术数据的挖掘使用。
5)与其它技术的联合:比如Crispr;
6)临床应用:按照这篇文章的观点,单细胞测序往临床上用的时间点大约在5年。
上一篇:实体组织制备温和组织处理器